中文/English
数据库

- OsPAD

OsPAD是一个水稻(Oryza sativa)系统蛋白质组注释数据库,包含基于二维聚丙烯酰胺凝胶电泳的集成化水稻蛋白质组学数据。OsPAD的目标是向生物学家提供一个水稻蛋白质组学信息的免费综合资源及其他一些在线工具。
Reference: Lili Liu, Lin Bai, Cong Luo, Donglin Huang, Ming Chen* (2011) Systematic Annotation and Bioinformatics Analyses of Large-scale Oryza sativa Proteome. Current Protein & Peptide Science, 12(7): 621-630.
- PmiRKB
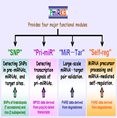
微型RNA(miRNAs),植物的一种小型RNA(sRNAs),在基因调控中扮演了重要角色。目前已建立了一些miRNA数据库;然而,需要收集、整理并分析连续生成的新数据。为了达到这一目标,我们建立了一个植物miRNA知识库(PmiRKB),它提供4个主要功能模块。在SNP模块中,收集了7个拟南芥(Arabidopsis thaliana)登记变种(accessions)和21个水稻(Oryza sativa)亚种的单核苷酸多态性数据,以检查pre-miRNA(前体微型RNA)和miRNA-目标的双链RNA中的SNP。根据其所在位置,SNP可能影响pre-miRNA的二级结构,或miRNA和靶分子间的作用。第二个模块,Pri-miR,可用在基于对RNA末端(PARE)数据分析研究组织特异性pre-miRNA和pri-miRNA(初级miRNA)的转录上下文。第三个模块,MiR-Tar,被设计用于通过对RNA末端(PARE)数据的平行分析验证数千个miRNA-靶分子对。相应的,第四个模块,Self-reg,同样使用PARE数据研究miRNA前体的代谢过程,包括前体处理及miRNA或miRNA*介导的对其宿主前体的自我调控效应。PmiRKB>可以通过http://10.71.115.115/pmirkb/免费访问。
Reference: Yijun Meng, Lingfeng Gou, Dijun Chen, Chuanzao Mao, Yongfeng Jin, Ping Wu, and Ming Chen* (2011) PmiRKB: a plant microRNA knowledge base. Nucleic Acids Research, 39(Database issue): D181-D187.
- PRIN
PRIN(水稻互作预测网络),是重要的模式生物——水稻的第一个高度注释的蛋白质交互数据库。它通过回归计算大幅度扩展了水稻目前已有的蛋白质-蛋白质交互数据,无疑将会为水稻功能基因组和系统生物学提供未来的展望。PRIN的蛋白质-蛋白质交互数据基于已进行大规模蛋白质-蛋白质交互实验的六种模式生物的interolog结果:酵母(Saccharomyces cerevisiae)、线虫(Caenorhabditis elegans)、果蝇(Drosophila melanogaster)、人(Homo Sapiens)、大肠杆菌(Escherichia coli)K12和拟南芥(Arabidopsis thaliana)。在一定质量的控制之下,我们总共获得了5049种水稻蛋白的76585项非冗余水稻蛋白质互作对。进一步的分析显示,预测的水稻蛋白质互作网络的拓补属性相比其余5种生物,更接近酵母的蛋白质互作网络。考虑到酵母的interolog贡献了全部互作的近74%,这个结果可能并不令人惊讶。此外,GO注释、亚细胞定位信息和基因表达数据同样以图谱形式存储于我们的网络上以供认证。最后,我们开发了一个对用户友好的网络界面,为数据库搜索和网络的可视化提供便利。
Reference: Haibin Gu, Pengcheng Zhu, Yinming Jiao, Yijun Meng and Ming Chen* (2011) PRIN, a predicted rice interactome network. BMC Bioinformatics, 12: 161.
- PlantNATsDB

自然反义转录子(NATs),作为调控RNA的一种,在植物基因组中普遍存在,并在生理和病理过程中扮演了重要角色。尽管NAT的生物功能已被广泛报道,但至今仍缺少一个综合的数据库。我们建立了一个涵盖69种植物约200万NAT对的植物NAT数据库(PlantNATsDB)。目前可用的GO注释和高通量小RNA测序数据均已集成到此数据库中以供研究NAT的生物功能。PlangNATsDB提供可变的对用户友好的网页界面以简化NAT的提交,此外还提供集成的图形化网络浏览器以显示不同NAT组成的复合网络。在此之外,还设计了一个基于GO注释的“基因集分析”模块以找出特定NAT网络的统计意义明显的超额GO范围。PlantNATsDB目前是植物界最为综合的NAT资源,可作为参考数据库为提供研究NAT调控功能提供服务。PlantNATsDB可以在http://bis.zju.edu.cn/pnatdb/免费访问。
Reference: Dijun Chen, Chun-hui Yuan, Jian Zhang, Zhao Zhang, Lin Bai, Yijun Meng, Ling-ling Chen*, Ming Chen* (2011) PlantNATsDB: a comprehensive database of plant natural antisense transcripts. Nucleic Acids Research, 40(Database issue): D1187-D1193.
- OsCAS

OsCAS(水稻芯片注释系统)是一个用于注释水稻微芯片实验结果及根据基因表达结果分析基因间关系的综合系统。此平台的目的在于在系统生物研究的框架内,简化微芯片实验中对不同表达基因的研究。OsCAS通过一个对用户友好的网页界面接受可用于通过用户选定的程序获取相关注释的基因芯片ID集合。目前这个系统集成了包括GeneBank、UniGene、SwissProt、TIGR、KOME、Kegg、Gene Ontology、MirBase等公共数据库,涵盖水稻的基因信息、蛋白质功能、代谢途径、调控因子等内容。OsCAS已被成功用于根据一些水稻微芯片实验结果注释大量的基因芯片ID,并简化多种基因的进一步分析过程。
Reference: Qingyun Shi, Yijun Meng, Dijun Chen, Fei He, Haibin Gu, Ping Wu, Ming Chen* (2010) OsCAS: a comprehensive web-based annotation platform for rice microarray data. BioChip Journal, 4(1): 9-15.
-
BioChip

我们开发了一个基于网页的平台,以提供高通量微芯片数据分析的网络服务程序。实施聚焦于微芯片数据的大量网络服务,也就是多任务和通过程序自动分析访问,而非手动与网页服务器互动。它包含一个提供大量探针和基因功能信息的集成注释系统。此外还有一些用于基因功能分类、基因集富集分析、协同表达发现、路径推断及弹性更强的模型建构的有用组件。
Reference: CHEN DiJun, ZHANG Fan, YUAN ChunHui, LU Jing, LI Xia, CHEN Ming* (2010) A web-based platform for rice microarray annotation and data analysis. Science in China Series C: Life Sciences, 53(12): 1459-1466.
欢迎对页面内容的评论和改进建议!
管理员:陈迪俊 © 2010-2015